Proposition de sujet de stage M2/PFE Ingénieur 2025-2026
Le stage a pour objectif de combiner les représentations neuronales implicites et les réseaux de neurones informés par la physique afin de modéliser les déformations d’organes alignées sur des données d’imagerie IRM, offrant ainsi une approche plus continue, précise et physiquement cohérente de la dynamique des organes observés.
Encadrement
Marc-Emmanuel Bellemare (Maître de conférences – HDR).
Christophe Roman (Maître de conférences).
marc-emmanuel.bellemare@lis-lab.fr ; christophe.roman@lis-lab.fr
Localisation
Laboratoire d’ Informatique et des Systèmes (LIS UMR 7020)
Domaine universitaire de Saint Jérôme, Avenue Escadrille Normandie Niemen, 13397 MARSEILLE Cedex 20.
https://www.lis-lab.fr
Description
Contexte
La modélisation des déformations des organes abdominaux revêt une importance cruciale pour la santé des patients et pour de nombreuses applications cliniques, telles que la planification de la radiothérapie adaptative, le suivi de la progression des maladies ou encore l’analyse biomécanique des tissus. L’imagerie par résonance ma- gnétique (IRM) peut offrir une visualisation spatiale et en coupe des déformations d’organes in vivo. Cependant, l’état de l’art actuel présente plusieurs limitations, notamment en termes de résolution et de reconstruction fidèle de l’évolution tridimensionnelle et dynamique des organes. L’objectif de ce stage est de proposer des solutions innovantes pour pallier ces limites.
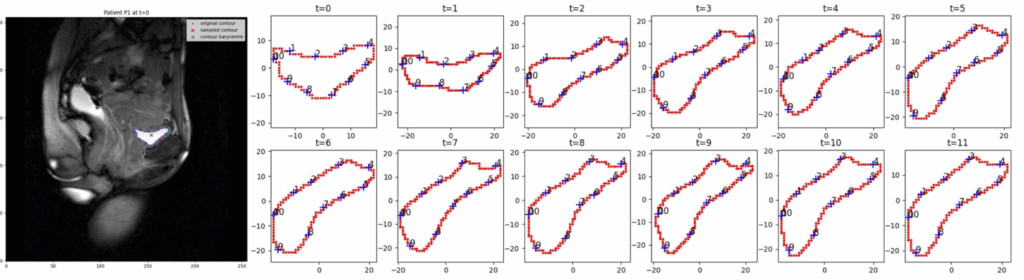
Dans le cadre d’un projet de recherche mené en collaboration avec l’AP-HM, nous nous intéressons au suivi des déformations des principaux organes pelviens. L’approche actuelle [1, 4] consiste à détecter un contour sur une série d’images 2D, puis à effectuer un échantillonnage spatial de ce contour initial. Les contours suivants sont ensuite estimés de manière récurrente à l’aide d’un modèle de transport optimal, la déformation finale étant calculée à partir de la distance entre les points d’échantillonnage obtenus (Figure 1). Cependant, cette méthode présente plusieurs faiblesses : la construction de l’échantillonnage est souvent arbitraire, le transport optimal peut introduire des biais difficiles à maîtriser, et la définition même de la distance utilisée reste discutable. Ces éléments limitent la robustesse et la généralisabilité de l’approche, malgré son intérêt scientifique certain.
Afin de dépasser ces limitations, ce stage vise à exploiter des modèles et méthodes récents capables d’apporter plus de cohérence et de précision à la modélisation des déformations. Le premier axe d’amélioration concerne la discrétisation : celle-ci peut être évitée grâce aux représentations neuronales implicites (Implicit Neural Representations, INRs). Ces dernières reposent sur le principe d’approximation universelle des réseaux de neurones, leur permettant de représenter n’importe quelle forme continue. Ainsi, le contour précédemment échantillonné sera alors directement modélisé par un réseau neuronal.
Pour l’estimation des déformations, nous proposons d’utiliser des réseaux de neurones informés par la physique (Physics-Informed Neural Networks, PINNs). L’idée est d’intégrer des contraintes issues des équations mécaniques de la déformation afin d’estimer à la fois le champ de déformation et les paramètres des lois de comportement des tissus.
Réalisation
L’objectif principal de ce stage est de développer et d’évaluer des modèles de déformation d’organes, en s’appuyant sur des approches d’apprentissage profond et de modélisation physique.
Les étapes et objectifs clés sont les suivants :
— Développer une représentation neuronale implicite (INR) des contours de déformation de la vessie en 2D + temps.
— Évaluer les performances de cette représentation en termes de précision et de continuité temporelle.
— Concevoir une approche basée sur les réseaux de neurones informés par la physique afin de reconstruire le champ de déformation et d’estimer les paramètres mécaniques du comportement.
— Évaluer les performances du PINN selon deux perspectives possibles :
– comme modèle hybride, intégrant à la fois les données expérimentales et les contraintes issues des équations physiques, afin de guider l’apprentissage vers des solutions cohérentes avec les lois mécaniques.
– ou comme problème inverse, visant à identifier les paramètres physiques (par ex. propriétés mécaniques des tissus) et les déformations spatiales à partir des données observées, tout en respectant les équations de la mécanique des milieux continus.
— Perspective d’extension vers la 3D+temps
Données
Le projet s’appuiera sur un jeu de données d’IRM dynamiques de la vessie, collecté auprès de 50 patientes. L’échantillonnage temporel, à raison d’une image par seconde, des séquences sagittales dynamiques fournit 12 images par patiente. Les contours de la vessie ont été extraits de manière manuelle ou semi-automatique, sur l’ensemble des images de la séquence dynamique. Au total, 600 contours ont ainsi été obtenus, constituant la base de données utilisée pour l’apprentissage et l’évaluation des performances du modèle.
Littérature associée
Une partie du stage sera également consacrée à une étude et une veille bibliographique. Le travail le plus proche de notre objectif est présenté dans [3], qui adopte une démarche très similaire à la nôtre. Les auteurs proposent une approche combinant une représentation neuronale implicite (INR) et un réseau de neurones informé par la physique (PINN), appliquée à une équation d’onde dans le but de reconstruire le coefficient d’élasticité spatial.
Dans [2], les auteurs s’intéressent au problème de propagation ainsi qu’au problème inverse associé aux équations des poutres. La partie consacrée au modèle de poutre de Timoshenko présente un intérêt particulier pour notre travail, car elle met en œuvre deux équations d’onde unidimensionnelles couplées.
Profils recherchés
Le ou la candidat.e sera intéressé.e par un domaine pluridisciplinaire embrassant l’analyse d’image, les mathématiques appliquées, le deep-learning, dans un contexte médical. Une expérience de la programmation avec l’environnement python est un pré-requis, la connaissance de la bibliothèque JAX serait un plus. Le stage aura une durée de 4 à 6 mois avec la gratification d’usage (de l’ordre de 600€ par mois).
Des connaissances en équations aux dérivées partielles (EDP) et en méthodes de résolution par éléments finis constituent un atout supplémentaire pour ce stage.
Candidature
Les candidats sont invités à transmettre leurs candidatures par mél aux encadrants. Le dossier de candidature comprendra un CV détaillé, une lettre de motivation pour le projet et les derniers relevés de notes.
Références
[1] Aivar, M. Etude des deformations 2D d’organes à tissus mous à l’aide d’IRM. Matser thesis, ENSE3(2023).
[2] Kapoor, T., Wang, H., Núñez, A., and Dollevoet, R. Physics-informed neural networks for solving forward and inverse problems in complex beam systems. IEEE Transactions on Neural Networks and Learning Systems 35, 5 (2023), 5981–5995.
[3] Kim, Y., Lee, J. Y., and Kwon, J. Physics-informed neural operators for tissue elasticity reconstruction. In Inter- national Conference on Medical Image Computing and Computer-Assisted Intervention (2025), Springer, pp. 381–390.
[4] Rahim, M., Bellemare, M.-E., Bulot, R., and Pirró, N. A diffeomorphic mapping based characterization of temporal sequences : application to the pelvic organ dynamics assessment. Journal of mathematical imaging and vision 47, 1 (2013), 151–164.